肺癌及其相关副发病变的遗传易感性
引言
肺癌在许多国家都是导致癌症死亡和疾病负担的首要原因[1,2]。尽管在肿瘤学的一些研究领域取得了巨大进步,但肺癌的治疗和预后仍没有明显改善。除了采取戒烟的策略外,通过了解肺癌发病中涉及的生物学通路来发现可能在预测方面、预后方面或诊断标志物或者新的治疗靶点等重要临床价值的关键生物分子是非常必要的。
与其他人类肿瘤一样,肺癌由一系列DNA序列或其表达异常导致。肺癌主要包括两类组织学亚型:非小细胞肺癌(non-small cell lung cancer,NSCLC)和小细胞肺癌(small cell lung cancer,SCLC)。NSCLC目前可根据发生在多个癌基因上的“驱动”突变来归类[3-5]。不过,由于与生殖细胞中的单核苷酸多态性(single nucleotide polymorphisms,SNPs)相关的肺癌不依赖于肿瘤发生的体细胞突变,研究者对其遗传易感性的研究仍然抱有很大兴趣[6,7]。
本综述将探讨肺癌的遗传易感性的最新观点。首先,概括遗传易感性的研究设计原则,特别是在过去至少5年内肺癌遗传学领域取得重大进展的全基因组相关性研究(Genome-wide association studies,GWAS)。其次,对已报道肺癌的GWAS以及迄今为止发现的候选易感基因进行说明。最后,总结针对吸烟行为和慢性阻塞性肺疾病(chronic obstructive pulmonary disease,COPD)易感性的GWAS,以作为最常见的导致肺癌(吸烟)和其慢性疾病危险因素(COPD)的相关遗传标记。
肺癌的遗传易感性——研究设计
许多研究已经明确了肺癌的遗传易感性,包括家族性风险分析[8]和独立分析的研究[9]。然而,遗传对肺癌的影响充其量是中等的。例如,Czene等通过采用960万个体的瑞典家庭癌症数据库估计遗传率仅为8%[10],在双胞胎研究中已经注意到异卵双胞胎比同卵双胞胎有更高的一致性[11]。吸烟是目前发现的致癌性最强的环境因素,这可能是与调节吸烟行为的基因[鉴于吸烟习惯的高遗传性,在双胞胎研究中为〜0.5[12]]而非那些决定癌变个体易感性的基因的遗传效应发挥着更重要的作用相关。而Lorenzo Bermejo等则推断吸烟对肺癌相对风险取决于吸烟者将吸烟习惯传递给后代的程度(吸烟遗传性)、一般人群的吸烟率及与非吸烟者相比吸烟者患肺癌的风险[13]。他们发现当以已发表吸烟行为的数据为蓝本时,吸烟对肺癌患者后代罹患肺癌相关风险为1.19,这提示肺癌的家族性病例不能归咎于共同的吸烟习惯。
由于肺癌有明显的遗传决定因子,可以利用许多其他的研究方法来确定疾病易感性的遗传决定因子。
连锁分析包括提出一个模型来解释在一个家系中观察到的表型和基因型的遗传模式[14]。当一个基因产生的表型性状及其周边标记共遗传时,连锁是显而易见的。相反,那些异常表型相关的标记则作为染色体的独立分类和在减数分裂过程中的杂交而被随机分布在受影响的家庭成员中。关联研究并不检测等位基因的遗传模式,相反,它们是基于在受影响和不受影响个体的人群组之间等位基因频率比较的病例对照研究。一个特定的等位基因如果与对照组相比发生在受影响个体之间有一个显著增高的频率,则被认为与性状相关。然后再通过与未受影响的人群相比在受影响人群中的等位基因的频率比来评估个体性状的比值比。关联研究的最大问题是选择一个合适的对照组与受影响的群体进行比较。对家庭成员的全基因组连锁分析发现了高渗透、低频率癌症易感性基因,如乳腺癌的BRCA1和BRCA2和大肠癌的APC基因。
对于肺癌,一些研究试图使用全基因组连锁方式来确定易感基因位点。然而,尽管一些易感基因潜在的基因位点已被确定,例如,与家族性肺、喉、咽癌相关联的染色体6q23-25[15]区域,但仍没有发现因果基因。随着后续GWAS的进行(见下文),研究者发现肺癌结果与COPD和肺功能[16]结果之间有相当多的重叠[16]。
GWAS
基于阵列的SNP基因分型技术和人类基因组单体型测绘[17]的进展提出了同时确定贯穿个体基因组数以百万计的SNPs的可能性,这使得研究者可以通过扩展关联研究设计来独立评估在整个基因组的关联。 GWAS已经彻底改变了复杂常见病的遗传因素研究[18,19]。对于200多个从常见病到生理测量如身高、BMI和生物测量如循环血脂水平的表型,GWAS都提供了在人类基因组数百个不同位点强有力的统计关联(www.gwascentral.org/)。GWAS现在有明确的研究方法包括严格的基因型鉴定、质量控制、人口分层(基因控制)和统计技术[20]。由于需要进行大量的统计测试,仔细利用Bonferroni和FDR多重检验校是必不可少的。在一个染色体位置SNPs的P值低于1%FDR集群定义为一个“极大关联”区,可将其作为第一候选基因区,以进一步采用次要结局指标、基因数据库搜索和精细定位分析检查来发现在其他队列/人群中的因果基因位点和拷贝。显示最强相关性的SNP是必然位点是不太可能的,因为SNPs被选择在基因组区域的其他变异仅提供最大的覆盖范围,而不是生物学功能。一旦确定候选区域或基因,可以在病例组和对照组之间以及不同基因型个体间比较基因表达差异,从而为该基因是否参与疾病提供进一步的证据。如果连锁不平衡阻止一个特定的基因的表达,那么可能需要利用不同种族和族裔群体以确定候选基因在GWAS所占遗传信号的比重[21]。
利用GWAS方法已发现许多有价值的肺癌易感基因,然而,阳性位点在家庭风险中仅占一个非常小的比例。使用GWAS方法确定常见疾病位点不能解释所有这些条件的遗传性(称为丢失的遗传性),这一发现也是目前很多争论的重点。许多可能存在的原因可以解释这一现象。其中包括基因—基因相互作用、基因—环境相互作用,以及其他类型的遗传变异,如罕见变异、结构变化和后生遗传等。将来可通过目前正在应用于其他复杂疾病的外显子或全基因组测序方法分析全基因组拷贝数变异和/或罕见变异,可能有助于进一步找出肺癌的遗传易感性位点[22]。然而,还有一种解决办法是利用新的分析方法来确定那些在采用GWAS方法中由于显著性水平要求严格而多重比较校正无法发现的弱相关变异。例如,在基因通路分析后利用一个比较宽松的多重修正,可以发现与疾病状态关联低于全基因组常规显著性水平的SNPs “灰色地带”参与常见生物信号通路的基因。使用类似的方法,Zhang等采用基因集富集分析(gene set enrichment analysis,GSEA)方法在汉族人群肺癌GWAS中进行了两个阶段的通路分析。研究发现四个通路(achPathway,metPathway,At1rPathway,rac1Pathway)中基因表达都有显著差异,这一结果可能会给肺癌病因提供新的线索[23]。
肺癌的GWAS
吸烟者肺癌易感性的GWAS
已经有许多GWAS在一定范围人群中进行,以测试肺癌易感性的的遗传影响(表1)。
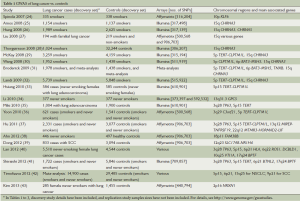
Full table
一项在意大利人群中进行的初步的、小规模GWAS报道显示了KLF6基因和SNPs的关联,但其结论不能在重复试验队列中验证[24]。另一项相对较小的家族性肺癌患者的研究则发现其与染色体15q区SNPs的关联[27]。在白种人群中至今已进行的研究有较大规模的GWAS,重复试验队列[25,26,28-30,32]和Meta分析[31,32,42]。这些GWAS发现肺癌风险与SNPs在统计学上有显著性关联,特别是在染色体15q,5p和的6p区(表1)。GWAS也已在亚洲肺癌人群中开展[35-37,39,41],并确定了一些与高加索人群研究中类似的SNPs,但是也发现了在亚洲人群中肺癌风险独特的其他SNPs[44]。
在许多研究中,观察到的与关键SNPs的关联是独立于吸烟状况或吸烟史的[25,26,29,32],尽管在某些研究中SNPs(如在15q)与吸烟行为有关[28]。在吸烟者研究中,与吸烟显著性的PAR(>80%)相比,计算出的这些SNPs的肺癌人群归因危险度(population attributable risk,PAR)是适度的,介于14%[26]和18%[28]之间。
特定肺癌人群的GWAS
GWAS也在不吸烟者中进行,并解决了其他一些与肺癌相关的问题。一项美国不吸烟肺癌患者的GWAS及重复试验队列发现了肺癌风险与在GPC5基因13q区的SNPs相关联[34]。该研究同时还检测了在不吸烟亚洲人群中的全基因组关联[38,40,43],以寻找新的不同于白种人群位点的易感基因位点。5p15区TERT基因的SNPs与特定的肺癌组织学类型肺腺癌有关[32],而9p21区的SNPs则与肺鳞状细胞癌(squamous cell carcinoma,SCC)相关[42]。肺癌风险和自我报告石棉暴露的GWAS也分析了基因—环境相互作用[45]。虽然这项试验研究没有发现显著性差异,但观察到了可能的基因—石棉暴露相互作用与7q32区C7orf54的SNPs相关联。此外,许多GWAS已经确定了可能预测SCLC[46]和NSCLC [47]患者化疗反应的SNPs,另外一些GWAS也发现了与肺癌患者预后相关的SNPs[48]。
GWAS肺癌易感性的候选基因
GWAS验证研究及其发现(表1)还有其他的一些验证研究已经确定了肺癌易感性的候选基因的显现模式[见综述[6,7,42]]。白种吸烟人群研究中结果一致的候选基因是15q25区的nAChR亚基(烟碱胆碱受体α3和5:CHRNA3和CHRNA5)。乙酰胆碱或尼古丁激活神经元nAChR,后者包括α和β亚基的亚聚体(五聚体)。nAChR在肺组织的神经元和非神经元细胞中表达,包括支气管上皮细胞和肺癌细胞。虽然nAChR的SNPs可能通过吸烟行为来改变患肺癌的风险,但这些SNPs还可以通过抗凋亡和增殖作用,或者烟雾中的尼古丁衍生致癌效应来调控尼古丁的直接影响(6,7,49-51 )。
在5p15位点,TERT和CLPTM1L基因SNPs在多项GWAS中发现与肺癌和其他癌症相关[52]。TERT基因编码人端粒酶逆转录酶,对维持端粒的长度非常重要。唇腭裂跨膜蛋白1样蛋白(cleft lip and palate transmembrane protein 1-like protein,CLPTM1L)可能在肺癌细胞中诱导细胞凋亡[29]。在TERT中发现的SNPs多为内含子,与在CLPTM1L中的SNPs连锁不平衡。6p21区的BAT3和MSH5已经在一些GWAS中发现其作为一种信号出现。BAT3(又名BAG6,BCL2-associated athanogene 6)编码参与DNA损伤诱导凋亡和p53基因毒性应激反应调制的一种核蛋白[7]。MSH5基因[MutS蛋白同源物(大肠埃希菌)] 则参与DNA错配修复。
虽然5p15区SNPs在高加索和亚洲人群重复研究中已经得到证实,但15q区的SNPs结果却不一致[40,53]。这种差异可能代表在吸烟和不吸烟人群中肺癌病因的不同(特别是在主要致癌物质可能为生物质燃料来源的室内空气污染的地方)。在不吸烟者,尤其是亚洲女性人群,也出现了其他一些候选基因,例如涉及受体酪氨酸激酶活性[ROS1,c-ROS癌基因1,受体酪氨酸激酶[40]],膜泡运输[VTI1A,膜泡运输作用于t-SNAREs 同源物1A[40]]和细胞黏附[NRXN1,neurexin1[43]]的基因。出乎意料的是转录因子TP63(tumor protein p63)也是不吸烟人群的候选标记。TP63编码一种蛋白质,它经常被用作为鳞状细胞癌(一种与吸烟者密切相关的癌症)的免疫组化标记[54,55]。
许多新发现的这些关联在肺癌易感性领域是全新的,在以前的肺癌候选基因研究中均未检出过,而之前这些研究则主要侧重于代谢酶[如CYP1A1[56-58]]、氧化应激途径和其他DNA修复机制等方面[7]。
GWAS和肺癌药理学
GWAS方法目前正在扩展用于研究肺癌的其他相关表型。例如,Han等最近开展了伊立替康联合顺铂化疗治疗非小细胞肺癌患者的生存期的GWAS,并确定了可以预测临床结果的候选SNPs[59]。
吸烟行为的GWAS
与肺癌病因相关的吸烟行为的GWAS已在大样本人群中开展(表2),而且集中在开始吸烟、吸烟量(每天吸烟数)和成功戒烟三个方面。研究者对肺癌易感性的兴趣不仅是与吸烟的因果关系,还有可能有直接生物效应的包括吸烟行为及肺癌常见的SNPs。
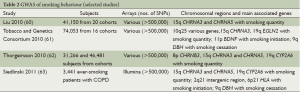
Full table
初次吸烟行为
研究已发现初次吸烟行为与11p区以及脑源性神经营养因子(brain-derived neurotrophic factor,BDNF)相关。神经妥乐平已作为一个可能的候选基因[61]。在一项COPD患者研究中发现与吸烟年龄相关的区域为6p21(HLA)和2q21(间隔区)[63]。
吸烟量
吸烟强度(吸烟量)在GWAS中均发现与15q25区SNPs一致相关[60-65],主要在nAChR基因位点(尤其是CHRNA3和CHRNA5)。其他涉及的位点包括19q区(CYP2A6,一种细胞色素P450尼古丁代谢酶)[61-63,66]和8p区(CHRNB3,nAChR亚基)[62]。
戒烟
一系列生物心理社会因素会影响一个人放弃吸烟的能力[67]。从GWAS中确定的戒烟的遗传因素主要在9q区的SNPs,包括参与多巴胺代谢的多巴胺β-羟化酶(dopamine beta-hydroxylase,DBH)[61,63]。
COPD的GWAS
肺癌和COPD通常在高危吸烟人群中同时存在。流行病学证据支持COPD的存在增加罹患肺癌风险的观点。除了烟雾,肺癌和COPD易感性的常见机制可能涉及了一系列生物过程,如炎症、上皮—间充质转换、修复异常、氧化应激和细胞增殖。此外,基因组和表观遗传学的变化可能改变生物途径,从而导致易患肺癌和COPD[68,69]。因此,研究COPD的遗传影响可以更好地发现肺癌和COPD共同的发病机制。重要的是,在对COPD和肺癌研究的设计和解释时应考虑遗传流行病学原则,因为两者之间存在共同的病因以及可能的遗传因素[70]。
COPD易感性的GWAS
在COPD中已经开展了许多GWAS研究(表3),但仍较肺癌的少。
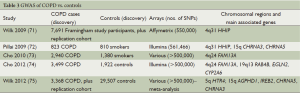
Full table
GWAS已经在Framingham(美国)队列[71]、Bergen(挪威)COPD队列(包括针对其他队列的重复研究)[72]、Bergen队列联合国家肺气肿治疗试验(National Emphysema Treatment Trial,NETT)以及COPD终点预测因子纵向评价(ECLIPSE)研究(Evaluation of COPD Longitudinally to Identify Predictive Surrogate Endpoints,ECLIPSE)[73]、4个合并队列[ECLIPSE、规范老龄化研究(Normative Aging Study)、Bergen(挪威)COPD队列和COPD基因][74]以及15个合并队列[75]研究中开展。
GWAS筛选的COPD候选基因
迄今从COPD的GWAS研究(表3)中已经发现了几个明显与COPD相关的区域。在15q位点(与肺癌和吸烟行为的GWAS类似)上,nAChR亚基基因(CHRNA5 CHRNA3)的SNPs与COPD相关[72,75],这可能提示COPD与作为肺癌其中一个病因的吸烟强度相关,尽管也应考虑到直接影响[76]。同样的,在19q13区的关联可能与吸烟行为相关[如CYP2A6[74]]。
在这些GWAS中也发现了其他新的与COPD关联的基因。在4q31区,Hedgehog相互作用蛋白(HHIP)在两项研究中被确定为候选基因[71,72]。HHIP编码的膜糖蛋白是参与COPD发展过程的Hedgehog信号通路的抑制剂。在BEAS-2B支气管上皮细胞株的基因表达研究中发现HHIP位于细胞外基质并与细胞增殖相关[77]。在4q24区,FAM13A(mily with sequence similarity 13, member A)在两项GWAS[73,74]中发现其SNPs与COPD和肺癌遗传相关联,提示两者可能存在共同的遗传途径[78]。FAM13A包含Rho GTP酶激活蛋白结合结构域,能够抑制信号转导和缺氧反应,但是其在肺脏的全部功能仍有待确定。在5q区,HTR4 [5-hydroxytryptamine (serotonin) receptor 4]在吸烟者中与COPD相关,其在呼吸道疾病的功能可能涉及调控细胞因子的释放[75]。
总结和临床意义
目前GWAS的主要发现
虽然肺癌主要是由吸烟引起的,但遗传易感性的作用在流行病学研究中已被广为认可。GWAS在肺癌以及吸烟行为和COPD的相关表型中已经完成。以白种人,吸烟者为主的大规模、多队列GWAS,已经确定染色体15q区(CHRNA3,CHRNA5)、5p区(TERT-CLPTM1L位点)和6p区(BAT3-MSH5)与肺癌风险强关联。一些在亚洲人群中对吸烟者的研究也发现了类似的风险位点,而其他一些GWAS尤其是在不吸烟的亚洲女性中,则确定了与吸烟相关的肺癌风险位点不同的其他风险位点。吸烟行为的GWAS发现与吸烟量相关的风险位点在15q(肺癌易感性的类似基因:CHRNA3,CHRNA5)和19q(CYP2A6)。其他基因则被定位为与开始吸烟和戒烟相关。在COPD,大样本GWAS也发现了定位在15q的风险位点NAChR SNPs,以及4q31(HHIP)、4q24(FAM13A)和5q(HTR4)等其他区域。肺癌、吸烟行为与COPD风险位点之间的重叠可能是由于尼古丁成瘾的影响,然而,需要进一步在基因—环境的相互作用及这些表型中探索尼古丁及其代谢产物的潜在直接影响。
应用和未来发展方向
从迄今为止的数据来看,GWAS不仅有助于阐明肺癌易感性的遗传影响,也有助于研究吸烟与肺癌因果关系的基因—环境相互作用,以及肺癌危险因素COPD的作用。肺癌及相关GWAS的结果可能在未来转化,使肺癌的个体风险、生物标志物诊断、预后和治疗标记的分析成为可能。现在面临的挑战是结合基因组学[79]、表观基因组学[80]、转录学[81-83]和蛋白质组学分析来改善肺癌患者和相关合并症的治疗[68]。未来的研究应包括肺癌和肺组织的DNA序列检测、基因组信息和临床表型的网络分析[3,84,85]。未来的肺癌遗传易感性研究的目标应着眼于在广泛肺癌人群中精炼最强的风险位点,并整合其他临床和生物标志物信息,通过增强诊断、预后、预防和治疗水平以达到对肺癌的个体化治疗[4]。
Acknowledgements
This work was supported by Asthma, Allergy & Inflammation Research (AAIR) (JWH), NHMRC Career Development Fellowship (IY), NHMRC Practitioner Fellowship (KF), and project grants from NHMRC, Queensland Health and Medical Research, The Prince Charles Hospital Foundation, and Cancer Council Queensland.
Disclosure: The authors declare no conflict of interest.
References
- Thun MJ, Lally CA, Flannery JT, et al. Cigarette smoking and changes in the histopathology of lung cancer. J Natl Cancer Inst 1997;89:1580-6. [PubMed]
- Mathers C, Vos T. The burden of disease and injury in australia-summary report. Canberra: Australian Institute of Health and Welfare, 1999.
- Sriram KB, Larsen JE, Yang IA, et al. Genomic medicine in non-small cell lung cancer: paving the path to personalized care. Respirology 2011;16:257-63. [PubMed]
- Fong KM, Yang IA, Bowman RV. Personalized medicine for lung cancer. Lung Cancer Management 2012;1:83-6.
- Sriram KB, Tan ME, Savarimuthu SM, et al. Screening for activating EGFR mutations in surgically resected nonsmall cell lung cancer. Eur Respir J 2011;38:903-10. [PubMed]
- Brennan P, Hainaut P, Boffetta P. Genetics of lung-cancer susceptibility. Lancet Oncol 2011;12:399-408. [PubMed]
- Marshall AL, Christiani DC. Genetic susceptibility to lung cancer--light at the end of the tunnel? Carcinogenesis 2013;34:487-502. [PubMed]
- Jonsson S, Thorsteinsdottir U, Gudbjartsson DF, et al. Familial risk of lung carcinoma in the Icelandic population. JAMA 2004;292:2977-83. [PubMed]
- Sellers TA, Chen PL, Potter JD, et al. Segregation analysis of smoking-associated malignancies: evidence for Mendelian inheritance. Am J Med Genet 1994;52:308-14. [PubMed]
- Czene K, Lichtenstein P, Hemminki K. Environmental and heritable causes of cancer among 9.6 million individuals in the Swedish Family-Cancer Database. Int J Cancer 2002;99:260-6. [PubMed]
- Braun MM, Caporaso NE, Page WF, et al. A cohort study of twins and cancer. Cancer Epidemiol Biomarkers Prev 1995;4:469-73. [PubMed]
- Tyndale RF. Genetics of alcohol and tobacco use in humans. Ann Med 2003;35:94-121. [PubMed]
- Lorenzo Bermejo J, Hemminki K. Familial lung cancer and aggregation of smoking habits: a simulation of the effect of shared environmental factors on the familial risk of cancer. Cancer Epidemiol Biomarkers Prev 2005;14:1738-40. [PubMed]
- Peddle L, Rahman P. Genetic epidemiology of complex phenotypes. Methods Mol Biol 2009;473:187-201. [PubMed]
- Bailey-Wilson JE, Amos CI, Pinney SM, et al. A major lung cancer susceptibility locus maps to chromosome 6q23-25. Am J Hum Genet 2004;75:460-74. [PubMed]
- Schwartz AG, Ruckdeschel JC. Familial lung cancer: genetic susceptibility and relationship to chronic obstructive pulmonary disease. Am J Respir Crit Care Med 2006;173:16-22. [PubMed]
- International HapMap Consortium. A haplotype map of the human genome. Nature 2005;437:1299-320. [PubMed]
- Altshuler D, Daly MJ, Lander ES. Genetic mapping in human disease. Science 2008;322:881-8. [PubMed]
- McCarthy MI, Abecasis GR, Cardon LR, et al. Genome-wide association studies for complex traits: consensus, uncertainty and challenges. Nat Rev Genet 2008;9:356-69. [PubMed]
- Amos CI. Successful design and conduct of genome-wide association studies. Hum Mol Genet 2007;16 Spec No. 2:R220-5.
- Lam AC, Powell J, Wei WH, et al. A combined strategy for quantitative trait loci detection by genome-wide association. BMC Proc 2009;3 Suppl 1:S6.
- Frazer KA, Murray SS, Schork NJ, et al. Human genetic variation and its contribution to complex traits. Nat Rev Genet 2009;10:241-51. [PubMed]
- Zhang R, Zhao Y, Chu M, et al. Pathway analysis for genome-wide association study of lung cancer in Han Chinese population. PLoS One 2013;8:e57763. [PubMed]
- Spinola M, Leoni VP, Galvan A, et al. Genome-wide single nucleotide polymorphism analysis of lung cancer risk detects the KLF6 gene. Cancer Lett 2007;251:311-6. [PubMed]
- Amos CI, Wu X, Broderick P, et al. Genome-wide association scan of tag SNPs identifies a susceptibility locus for lung cancer at 15q25.1. Nat Genet 2008;40:616-22. [PubMed]
- Hung RJ, McKay JD, Gaborieau V, et al. A susceptibility locus for lung cancer maps to nicotinic acetylcholine receptor subunit genes on 15q25. Nature 2008;452:633-7. [PubMed]
- Liu P, Vikis HG, Wang D, et al. Familial aggregation of common sequence variants on 15q24-25.1 in lung cancer. J Natl Cancer Inst 2008;100:1326-30. [PubMed]
- Thorgeirsson TE, Geller F, Sulem P, et al. A variant associated with nicotine dependence, lung cancer and peripheral arterial disease. Nature 2008;452:638-42. [PubMed]
- McKay JD, Hung RJ, Gaborieau V, et al. Lung cancer susceptibility locus at 5p15.33. Nat Genet 2008;40:1404-6. [PubMed]
- Wang Y, Broderick P, Webb E, et al. Common 5p15.33 and 6p21.33 variants influence lung cancer risk. Nat Genet 2008;40:1407-9. [PubMed]
- Broderick P, Wang Y, Vijayakrishnan J, et al. Deciphering the impact of common genetic variation on lung cancer risk: a genome-wide association study. Cancer Res 2009;69:6633-41. [PubMed]
- Landi MT, Chatterjee N, Yu K, et al. A genome-wide association study of lung cancer identifies a region of chromosome 5p15 associated with risk for adenocarcinoma. Am J Hum Genet 2009;85:679-91. [PubMed]
- Hsiung CA, Lan Q, Hong YC, et al. The 5p15.33 locus is associated with risk of lung adenocarcinoma in never-smoking females in Asia. PLoS Genet 2010;6:e1001051. [PubMed]
- Li Y, Sheu CC, Ye Y, et al. Genetic variants and risk of lung cancer in never smokers: a genome-wide association study. Lancet Oncol 2010;11:321-30. [PubMed]
- Miki D, Kubo M, Takahashi A, et al. Variation in Tp63 Is Associated with Lung Adenocarcinoma Susceptibility in Japanese and Korean Populations. Nat Genet 2010;42:893-6. [PubMed]
- Yoon KA, Park JH, Han J, et al. A genome-wide association study reveals susceptibility variants for non-small cell lung cancer in the Korean population. Hum Mol Genet 2010;19:4948-54. [PubMed]
- Hu Z, Wu C, Shi Y, et al. A genome-wide association study identifies two new lung cancer susceptibility loci at 13q12.12 and 22q12.2 in Han Chinese. Nat Genet 2011;43:792-6. [PubMed]
- Ahn MJ, Won HH, Lee J, et al. The 18p11.22 locus is associated with never smoker non-small cell lung cancer susceptibility in Korean populations. Hum Genet 2012;131:365-72. [PubMed]
- Dong J, Jin G, Wu C, et al. Genome-wide association study identifies a novel susceptibility locus at 12q23.1 for lung squamous cell carcinoma in han chinese. PLoS Genet 2013;9:e1003190. [PubMed]
- Lan Q, Hsiung CA, Matsuo K, et al. Genome-wide association analysis identifies new lung cancer susceptibility loci in never-smoking women in Asia. Nat Genet 2012;44:1330-5. [PubMed]
- Shiraishi K, Kunitoh H, Daigo Y, et al. A genome-wide association study identifies two new susceptibility loci for lung adenocarcinoma in the Japanese population. Nat Genet 2012;44:900-3. [PubMed]
- Timofeeva MN, Hung RJ, Rafnar T, et al. Influence of common genetic variation on lung cancer risk: meta-analysis of 14 900 cases and 29 485 controls. Hum Mol Genet 2012;21:4980-95. [PubMed]
- Kim JH, Park K, Yim SH, et al. Genome-wide association study of lung cancer in korean non-smoking women. J Korean Med Sci 2013;28:840-7. [PubMed]
- Dong J, Hu Z, Wu C, et al. Association analyses identify multiple new lung cancer susceptibility loci and their interactions with smoking in the Chinese population. Nat Genet 2012;44:895-9. [PubMed]
- Wei S, Wang LE, McHugh MK, et al. Genome-wide gene-environment interaction analysis for asbestos exposure in lung cancer susceptibility. Carcinogenesis 2012;33:1531-7. [PubMed]
- Wu C, Xu B, Yuan P, et al. Genome-wide examination of genetic variants associated with response to platinum-based chemotherapy in patients with small-cell lung cancer. Pharmacogenet Genomics 2010;20:389-95. [PubMed]
- Wu X, Ye Y, Rosell R, et al. Genome-wide association study of survival in non-small cell lung cancer patients receiving platinum-based chemotherapy. J Natl Cancer Inst 2011;103:817-25. [PubMed]
- Lee Y, Yoon KA, Joo J, et al. Prognostic implications of genetic variants in advanced non-small cell lung cancer: a genome-wide association study. Carcinogenesis 2013;34:307-13. [PubMed]
- West KA, Brognard J, Clark AS, et al. Rapid Akt activation by nicotine and a tobacco carcinogen modulates the phenotype of normal human airway epithelial cells. J Clin Invest 2003;111:81-90. [PubMed]
- Tournier JM, Birembaut P. Nicotinic acetylcholine receptors and predisposition to lung cancer. Curr Opin Oncol 2011;23:83-7. [PubMed]
- Al-Wadei HA, Al-Wadei MH, Schuller HM. Cooperative regulation of non-small cell lung carcinoma by nicotinic and beta-adrenergic receptors: a novel target for intervention. PLoS One 2012;7:e29915. [PubMed]
- Mocellin S, Verdi D, Pooley KA, et al. Telomerase reverse transcriptase locus polymorphisms and cancer risk: a field synopsis and meta-analysis. J Natl Cancer Inst 2012;104:840-54. [PubMed]
- Truong T, Hung RJ, Amos CI, et al. Replication of lung cancer susceptibility loci at chromosomes 15q25, 5p15, and 6p21: a pooled analysis from the International Lung Cancer Consortium. J Natl Cancer Inst 2010;102:959-71. [PubMed]
- Khayyata S, Yun S, Pasha T, et al. Value of P63 and CK5/6 in distinguishing squamous cell carcinoma from adenocarcinoma in lung fine-needle aspiration specimens. Diagn Cytopathol 2009;37:178-83. [PubMed]
- Zhang P, Han Y, Huang L, et al. Expression and Clinical Significance of TTF-1 and p63 in NSCLC. Zhongguo Fei Ai Za Zhi 2009;12:995-9. [PubMed]
- Larsen JE, Colosimo ML, Yang IA, et al. Risk of non-small cell lung cancer and the cytochrome P4501A1 Ile462Val polymorphism. Cancer Causes Control 2005;16:579-85. [PubMed]
- Larsen JE, Colosimo ML, Yang IA, et al. CYP1A1 Ile462Val and MPO G-463A interact to increase risk of adenocarcinoma but not squamous cell carcinoma of the lung. Carcinogenesis 2006;27:525-32. [PubMed]
- Wright CM, Larsen JE, Colosimo ML, et al. Genetic association study of CYP1A1 polymorphisms identifies risk haplotypes in nonsmall cell lung cancer. Eur Respir J 2010;35:152-9. [PubMed]
- Han JY, Lee YS, Shin ES, et al. A genome-wide association study of survival in small-cell lung cancer patients treated with irinotecan plus cisplatin chemotherapy. Pharmacogenomics J 2013. [Epub ahead of print]. [PubMed]
- Liu JZ, Tozzi F, Waterworth DM, et al. Meta-analysis and imputation refines the association of 15q25 with smoking quantity. Nat Genet 2010;42:436-40. [PubMed]
- Tobacco and Genetics Consortium. Genome-wide meta-analyses identify multiple loci associated with smoking behavior. Nat Genet 2010;42:441-7. [PubMed]
- Thorgeirsson TE, Gudbjartsson DF, Surakka I, et al. Sequence variants at CHRNB3-CHRNA6 and CYP2A6 affect smoking behavior. Nat Genet 2010;42:448-53. [PubMed]
- Siedlinski M, Cho MH, Bakke P, et al. Genome-wide association study of smoking behaviours in patients with COPD. Thorax 2011;66:894-902. [PubMed]
- Saccone NL, Culverhouse RC, Schwantes-An TH, et al. Multiple independent loci at chromosome 15q25.1 affect smoking quantity: a meta-analysis and comparison with lung cancer and COPD. PLoS Genet 2010;6:e1001053. [PubMed]
- David SP, Hamidovic A, Chen GK, et al. Genome-wide meta-analyses of smoking behaviors in African Americans. Transl Psychiatry 2012;2:e119. [PubMed]
- Kumasaka N, Aoki M, Okada Y, et al. Haplotypes with copy number and single nucleotide polymorphisms in CYP2A6 locus are associated with smoking quantity in a Japanese population. PLoS One 2012;7:e44507. [PubMed]
- Fung PR, Snape-Jenkinson SL, Godfrey MT, et al. Effectiveness of hospital-based smoking cessation. Chest 2005;128:216-23. [PubMed]
- Yang IA, Relan V, Wright CM, et al. Common pathogenic mechanisms and pathways in the development of COPD and lung cancer. Expert Opin Ther Targets 2011;15:439-56. [PubMed]
- Houghton AM. Mechanistic links between COPD and lung cancer. Nat Rev Cancer 2013;13:233-45. [PubMed]
- El-Zein RA, Young RP, Hopkins RJ, et al. Genetic predisposition to chronic obstructive pulmonary disease and/or lung cancer: important considerations when evaluating risk. Cancer Prev Res (Phila) 2012;5:522-7. [PubMed]
- Wilk JB, Chen TH, Gottlieb DJ, et al. A genome-wide association study of pulmonary function measures in the Framingham Heart Study. PLoS Genet 2009;5:e1000429. [PubMed]
- Pillai SG, Ge D, Zhu G, et al. A genome-wide association study in chronic obstructive pulmonary disease (COPD): identification of two major susceptibility loci. PLoS Genet 2009;5:e1000421. [PubMed]
- Cho MH, Boutaoui N, Klanderman BJ, et al. Variants in FAM13A are associated with chronic obstructive pulmonary disease. Nat Genet 2010;42:200-2. [PubMed]
- Cho MH, Castaldi PJ, Wan ES, et al. A genome-wide association study of COPD identifies a susceptibility locus on chromosome 19q13. Hum Mol Genet 2012;21:947-57. [PubMed]
- Wilk JB, Shrine NR, Loehr LR, et al. Genome-wide association studies identify CHRNA5/3 and HTR4 in the development of airflow obstruction. Am J Respir Crit Care Med 2012;186:622-32. [PubMed]
- Bossé Y. Updates on the COPD gene list. Int J Chron Obstruct Pulmon Dis 2012;7:607-31. [PubMed]
- Zhou X, Qiu W, Sathirapongsasuti JF, et al. Gene expression analysis uncovers novel hedgehog interacting protein (HHIP) effects in human bronchial epithelial cells. Genomics 2013;101:263-72. [PubMed]
- Young RP, Hopkins RJ, Hay BA, et al. FAM13A locus in COPD is independently associated with lung cancer - evidence of a molecular genetic link between COPD and lung cancer. Appl Clin Genet 2010;4:1-10. [PubMed]
- Sriram KB, Larsen JE, Savarimuthu Francis SM, et al. Array-comparative genomic hybridization reveals loss of SOCS6 is associated with poor prognosis in primary lung squamous cell carcinoma. PLoS One 2012;7:e30398. [PubMed]
- Davidson MR, Larsen JE, Yang IA, et al. Microrna-218 Is Deleted and Downregulated in Lung Squamous Cell Carcinoma. PLoS ONE 2010;5:e12560. [PubMed]
- Larsen JE, Pavey SJ, Bowman R, et al. Gene expression of lung squamous cell carcinoma reflects mode of lymph node involvement. Eur Respir J 2007;30:21-5. [PubMed]
- Larsen JE, Pavey SJ, Passmore LH, et al. Expression profiling defines a recurrence signature in lung squamous cell carcinoma. Carcinogenesis 2007;28:760-6. [PubMed]
- Larsen JE, Pavey SJ, Passmore LH, et al. Gene expression signature predicts recurrence in lung adenocarcinoma. Clin Cancer Res 2007;13:2946-54. [PubMed]
- Daniels M, Goh F, Wright CM, et al. Whole genome sequencing for lung cancer. J Thorac Dis 2012;4:155-63. [PubMed]
- Silverman EK. Perspective: how can genetics help? Nature 2012;489:S7. [PubMed]
(译者:林振宇;校对:骆华春)
(本译文仅供学术交流,实际内容请以英文原文为准。)


